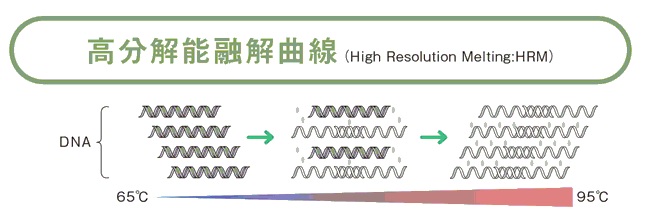
今回の識別法は、高分解能融解曲線分析(HRM解析法)という手法を用いる。
まず、ウイルスのゲノム(全遺伝情報)から、試薬(蛍光色素)を結合させたDNA二本鎖をPCRで作成する。
徐々に温度を上げていくと、二本鎖が一本ずつに分かれていき、その際に蛍光強度が変化する。
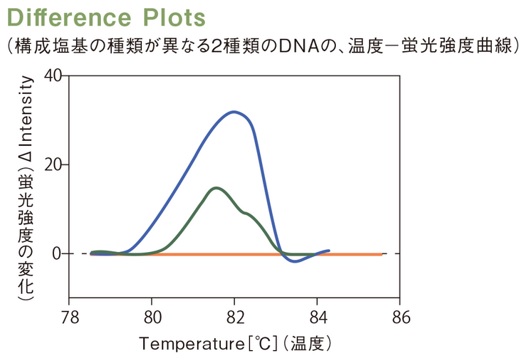
DNAを構成する塩基の種類が1個でも違えば、温度-蛍光強度グラフの曲線が異なってくる。その曲線の違いから変異の有無と変異株の種類が識別できる。
DNA二本鎖に蛍光色素を結合させておくと、1本ずつに分かれていく際に蛍光が減弱する。
蛍光の強度と温度の関係を示すグラフの曲線は、DNAを構成する塩基によって変る。
つまり、曲線が異なれば「遺伝子変異あり」と判別できる。予め各変異型の曲線が分かっているため、変異株の種類も識別できる。
ウイルスの実物がなくても、文献やデータベースにあるゲノム配列情報に基づいてDNAを合成する技術を導入することで解決 し、従来型、英国型、南アフリカ型、ブラジル型、フィリピン型、日本固有型、カリフォルニア型、インド型の8種類を識別できるようになった。
愛知県衛生研究所との実証実験は2021年4月に3週間にわたって行い、実物の209の陽性検体を用いて、従来型と英国型をほぼ正確に識別できた。
これまでは検体を国立感染症研究所に送り、およそ3万個の遺伝子すべてを解析するため1週間かか る。
この技術は遺伝子の一部の解析で済み、一般的なPCR検査の装置をそのまま使用して、いずれの変異株も90分程度で識別することが可能で、ゲノム解析に要する期間を大幅に短縮できる。
ーーー
神野教授は薬学部の実習で、名古屋コーチンなど鶏肉の銘柄を遺伝子の配列の違いから見分ける解析法を教えている。
「同じニワトリでも遺伝子の配列が違うので銘柄の違いが分かる。新型コロナウイルス(の従来株と変異株)も同様」と考えた。
新型コロナの変異株で試しても、種類によって光が弱くなる温度が異なることが分かった。
HRM解析法自体は一般的な手法だが、ゲノム増幅に用いるDNAの設計開発には同研究室独自の工夫があり、それが今回の研究にも生かされ た。
この研究は、衛生薬学分野を専門とし、日頃から社会貢献を意識して研究に臨んできた同研究室の教員・大学院生の「コロナ禍に対して何か貢献できないか?」という議論からスタートした。
今後新たな変異型の存在が報告された場合も、感染が拡大する前に、配列情報からDNAを合成して変異型の温度―蛍光強度特性を解析し、検出体制を速やかに整えられる。
このプロジェクトには、生物物理化学を専門とする薬学部の小田彰史教授も参加し、遺伝子変異が感染力や毒性に及ぼす影響をコンピューターで予測する方法を開発している。
新たな変異型が生じた場合、速やかに感染力や有害性を予測し、その型に応じた検出技術を確立するとしている。
ーーーーーーーーーーーーーー
前日以前のブログを続けてみる場合は、ページトップのタイトル(下の部分)をクリックしてください。
過去のデータは下記から利用できます。
ブログ & データベース専用検索
COVID-19関連はここをクリック
コメントする